grrr Julia ![]()
Dans python tu peux faire import julia. Ça marche très bien.
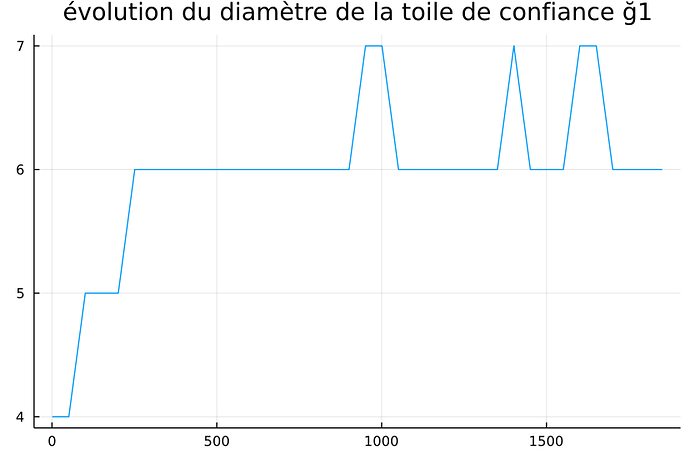
Je suis entrain d’étudier l’évolution de la toile G1 et je m’aperçois que le diamètre du graphe représentant la toile chute brusquement vers le 1400 IIème jours(en 2021). C’est comme si il y avait eu des conseils selon quoi il fallait détendre la toile en certifiant davantage les anciens membres de la toile.
Le diamètre d’un graphe c’est le plus long chemin parmi toute les distance minimum d’un sommet a un autre.
Est-ce que vous pouvez me confirmer cette hypothèse ?
Voici le graphe que j’ai:
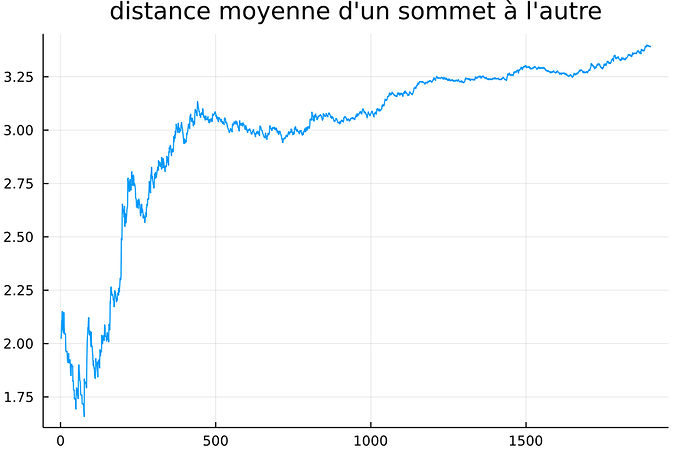
J’ai aussi ce graphe qui présente l’évolution (pour chaque jours) de la distance moyenne d’un sommet a un autre de la toile de confiance G1:
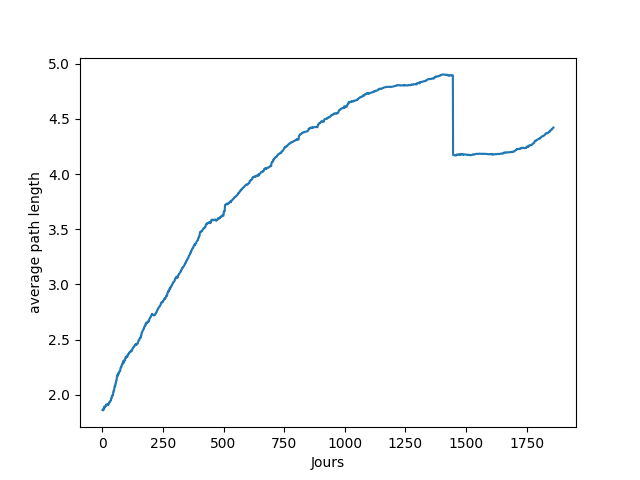
Je soupçonne que cette chute correspond plutôt à la chute du nombre de référents suite au palier des 3125 membres, mais je peux me tromper… Voir ce message :
Je suppose que tu parles du diamètre du graphe non dirigé comptant toutes les identités reliées par des certifications actives indépendamment de leur statut de membre. Peux-tu confirmer ?
Ce changement me paraît trop brusque pour une hypothèse impliquant une action humaine. Mais avant de me risquer sur des interprétations, j’aimerais bien vérifier le résultat. Je vais essayer de le faire ce soir avec DataJune. Est-ce que tu peux publier ton code aussi pour pouvoir comparer ?
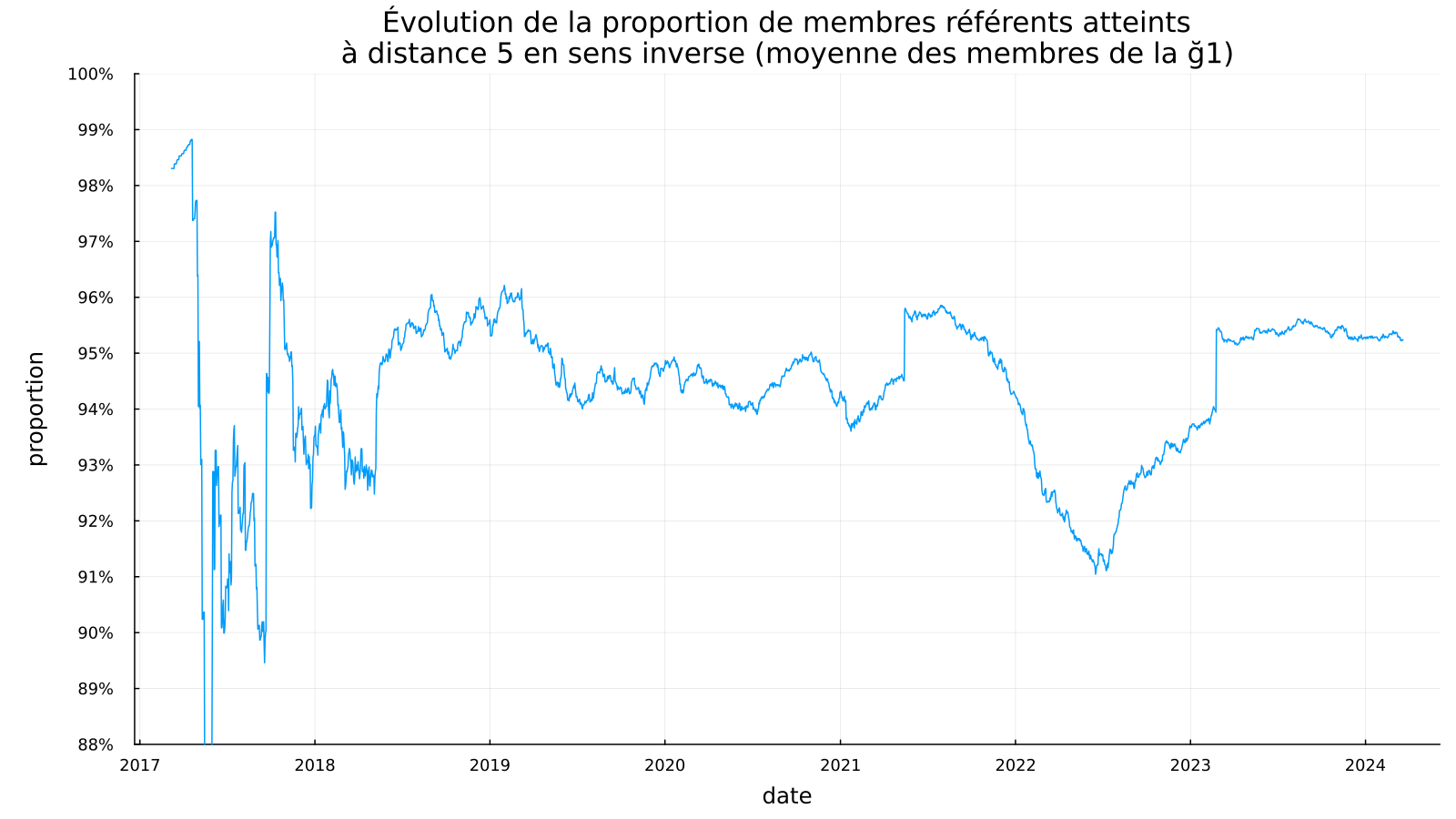
Le saut évoqué par @vit à propos des membres référents est visible sur ce graphe :
Mais comme tu regardes le diamètre du graphe indépendamment du critère « référent », l’hypothèse ne passe pas.
Est-ce que cela pourrais être dû au ralentissement de l’entrée de nouveaux membres à cause de la baisse du nombre de rencontres suite aux restrictions covid ?
Du coup certains en aurais profité pour certifier leurs connaissances déjà membres.
Mais si c’est sur une seule journée, ça ne tient pas mon idée.
Je parle du diamètre du graphe orienté, c’est a dire de la longueur maximum du plus petit chemin orienté pour toute combinaison de deux sommets du graphe.
Par exemple le diamètre de ce graphe:
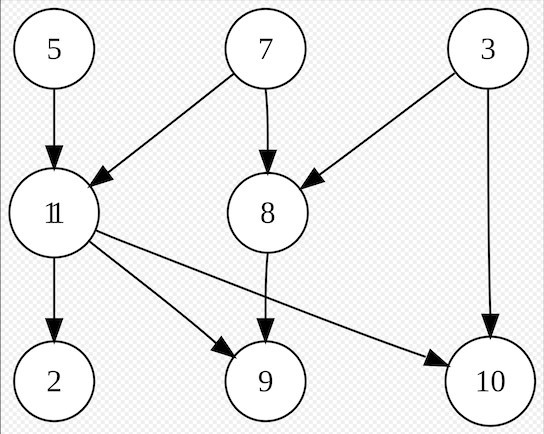
vaut 2, le plus long chemin entre n’importe quelle combinaison de deux sommets est de 2.
Je vais faire un dépôt gitlab ou je vais mettre mon code.
Sur ton exemple de graphe (tiré de Topological sorting - Wikipedia), si tu prends les noeuds 2 et 3, il n’y a pas de chemin orienté entre les deux. Donc comme le chemin n’existe pas, sa « longueur » est infinie. Je recommande de travailler sur le graphe non dirigé, ça assure de travailler sur un graphe connexe et je trouve que ça a plus de sens étant donné qu’une certification A→B veut dire « A a rencontré B en personne », et donc c’est censé être réciproque.
Ok, mais ca n’explique quand même pas la chute brusque il me semble.
Le depot va se trouver ici ; Antoine Millon / Duniter-analysis · GitLab
Avant de chercher à expliquer un phénomène, c’est bien de vérifier ce qu’on observe vraiment. Par exemple :
julia> using Graphs
julia> g = SimpleDiGraph(2)
{2, 0} directed simple Int64 graph
julia> add_edge!(g, 1,2)
true
julia> diameter(g)
┌ Warning: Infinite path length detected for vertex 2
└ @ Graphs ~/.julia/packages/Graphs/zrMoC/src/distance.jl:74
9223372036854775807
Dans ce cas, ma définition du diamètre du graphe me donne une valeur infinie, et donc ici la valeur maximale représentée par Int64. Chercher à interpréter ça alors qu’il y a un problème de définition n’a pas de sens.
Merci pour le dépôt. Je regarderai ^^
(et n’hésite pas à utiliser un fichier .gitignore pour exclure certains fichiers de ton dépôt)
Ma librairie ne renvoie pas une valeur infinie lorsqu’il n’existe pas de chemin orienté entre deux sommets. Il ne prend simplement pas en compte le chemin. J’ai vérifié et ca reste cohérent. Bon bref je vais modifier le code pour qu’il prenne en compte un graphe non orienté.
PS: je suis pas très habitué a utiliser git
Mon depot git n’est pas clair mais je vais le clarifier demain.
En quelques lignes de DataJune :
Je produis le graphe suivant (un point tous les cinquante jours) :
C’est pour le graphe non dirigé, donc c’est normal que le diamètre soit plus faible, mais je ne vois pas la même discontinuité que toi.
[edit]
et là j’ai calculé la distance moyenne pour un graphe dirigé en excluant les valeurs infinies :
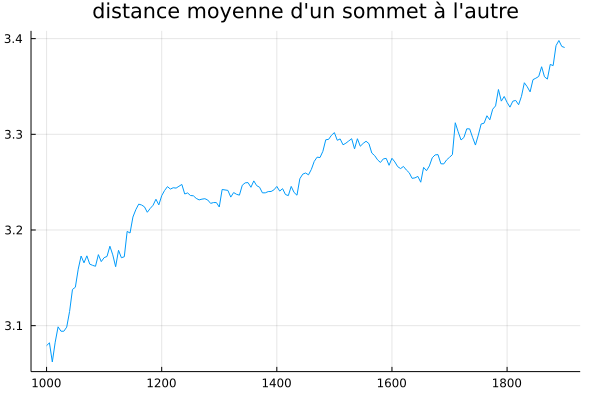
entre 1000 et 1900 avec un pas de 5 jours
les valeurs ne sont pas les mêmes que toi : je suis entre 3.1 et 3.4 alors que tu es entre 4.0 et 5.0
et comme je n’ai pas de discontinuité j’ai tendance à penser que mon résultat est le bon ![]()
je vais regarder ton code
[edit]
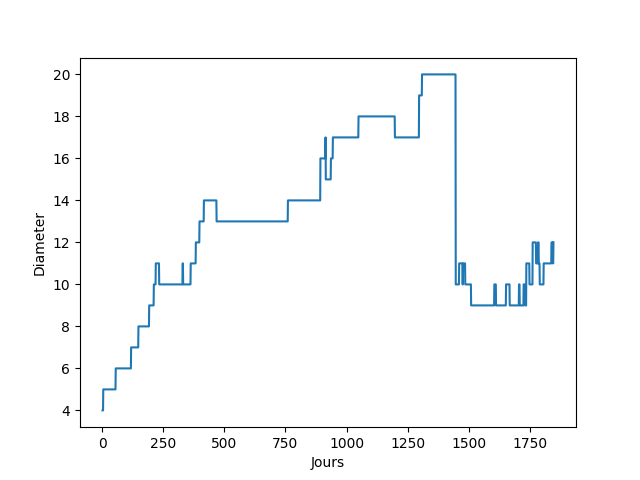
ça a mis une demi heure à calculer parce que j’y suis allé comme une brutasse, mais voilà le résultat que j’obtiens avec un pas de 1 jour :
pas du tout la même forme que toi. Voici le fichier pour que tu puisses comparer avec ton résultat :
mean_distance.txt (34.4 KB)
et le code s’il prenait l’envie à quelqu’un d’essayer DataJune :
using DataJune.WotHistory
using Graphs, Plots, ProgressMeter, StatsBase
w = WotHistory.WotSnapshot()
steps = 1:1902
mean_i = [] # mean distance for each step
@showprogress "Computing" for i in steps
WotHistory.set_current_interval!(w, i)
mean_j = [] # mean distance for each node
for j in 1:nv(w.g)
# tous les membres (même ceux dont l'adhésion a expiré par exemple)
distances = gdistances(w.g, j) # compute geodesic distances
m = mean(distances[distances .< typemax(Int64)]) # distance moyenne parmi les distances finies
push!(mean_j, m)
end
push!(mean_i, mean(mean_j))
end
plot(steps, mean_i, label=false)
title!("distance moyenne d'un sommet à l'autre")
(avant ça il faut faire :
DataJune.BlockchainBrowser.sync()
DataJune.BlockchainBrowser.browse()
DataJune.LightGraphExporter.lightgraph_export()
pour récupérer les données comme précisé dans le README)
C’est assez clair pour voir que tu utilises la méthode closeness dont la définition est :
https://igraph.org/python/api/latest/igraph._igraph.GraphBase.html#closeness
def closeness(vertices=None, mode='all', cutoff=None, weights=None, normalized=True):Calculates the closeness centralities of given vertices in a graph.
The closeness centerality of a vertex measures how easily other vertices can be reached from it (or the other way: how easily it can be reached from the other vertices). It is defined as the number of vertices minus one divided by the sum of the lengths of all geodesics from/to the given vertex.
If the graph is not connected, and there is no path between two vertices, the number of vertices is used instead the length of the geodesic. This is always longer than the longest possible geodesic.
Donc on ne parle pas exactement de la même chose.
À voir l’explication de la discontinuité dans le cas de la closeness… Mais là ![]()
Merci beaucoup! Je crois savoir qu’elle est mon erreur. Au sujet de la closeness, le code que j’ai mis sur git est pas celui qui me donner les résultats que j’ai montré, j’utilisais la methode ‹ diameter › de igraph pour me donner le graphique au sujet de l’évolution du diamètre. Je vais essayer de corriger l’erreur.
La closeness se calcule au niveau de chaque sommet du graphe, elle correspond a une valeur numérique représentant a qu’elle point le sommet est loin de tout les autres (en terme de longueur du chemin minimum). Mon idée a propos de la closeness etait la suivante: afficher une surface 3D representant l’evolution de la closeness de chaque sommet pour chaque graphe (jour après jour).
Si on etudie les 8000 sommets on voit pas grand chose pour l’instant, donc j’affiche la closeness que pour les 10 sommets les plus anciens:
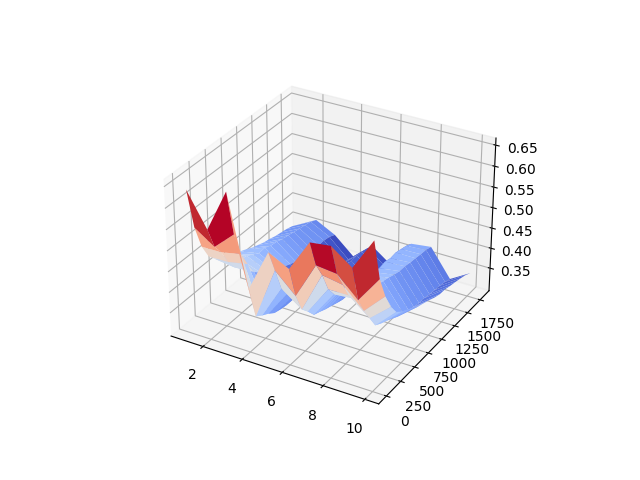
L’abscisse de 1 a 10 représente les differents sommets etudié, l’abscisse de 0 a 1750 représente les différents jours et la hauteur représente la valeur de la closeness. Plus celle-ci est grande plus le sommet est ‘proche’ de tout les autres en moyenne.
Globalement on remarque que plus la toile grandit moins les sommets sont proche de tout les autres.
Bon j’ai modifié le dépôt mais pas encore eu le temps de trouver l’erreur, mais je suis presque sur que ca doit être liée au fait qu’au milieu de la conversion des fichiers .lg en .pickle j’avais modifié l’orientation du graphe (de undirected a directed). Demain je répare mes donnees.
@HugoTrentesaux Est-ce que dans les fichiers .lg les sommets sont identifié? Je veux dire est-ce que le sommet numero 1 dans le fichier 000.lg est le même que le sommet numero 1 dans le fichier 1900.lg par exemple?